南湖新闻网讯(通讯员 樊亚伟、张俊成、张德建)2021年9月28日,Nature Communications在线发表了华中农业大学作物遗传改良国家重点实验室水稻团队、湖北洪山实验室李一博教授课题组题为“The identification of grain size genes by RapMap reveals directional selection during rice domestication”的研究论文。该研究开发了一种快速、高通量克隆数量性状位点(QTL)基因的新方法——RapMap(图1),发现了单位点遗传的本质判断标准,快速克隆了8个水稻种子大小QTL基因,并揭示了籼稻细长粒形是在水稻长期驯化和改良进程中定向选择的结果。
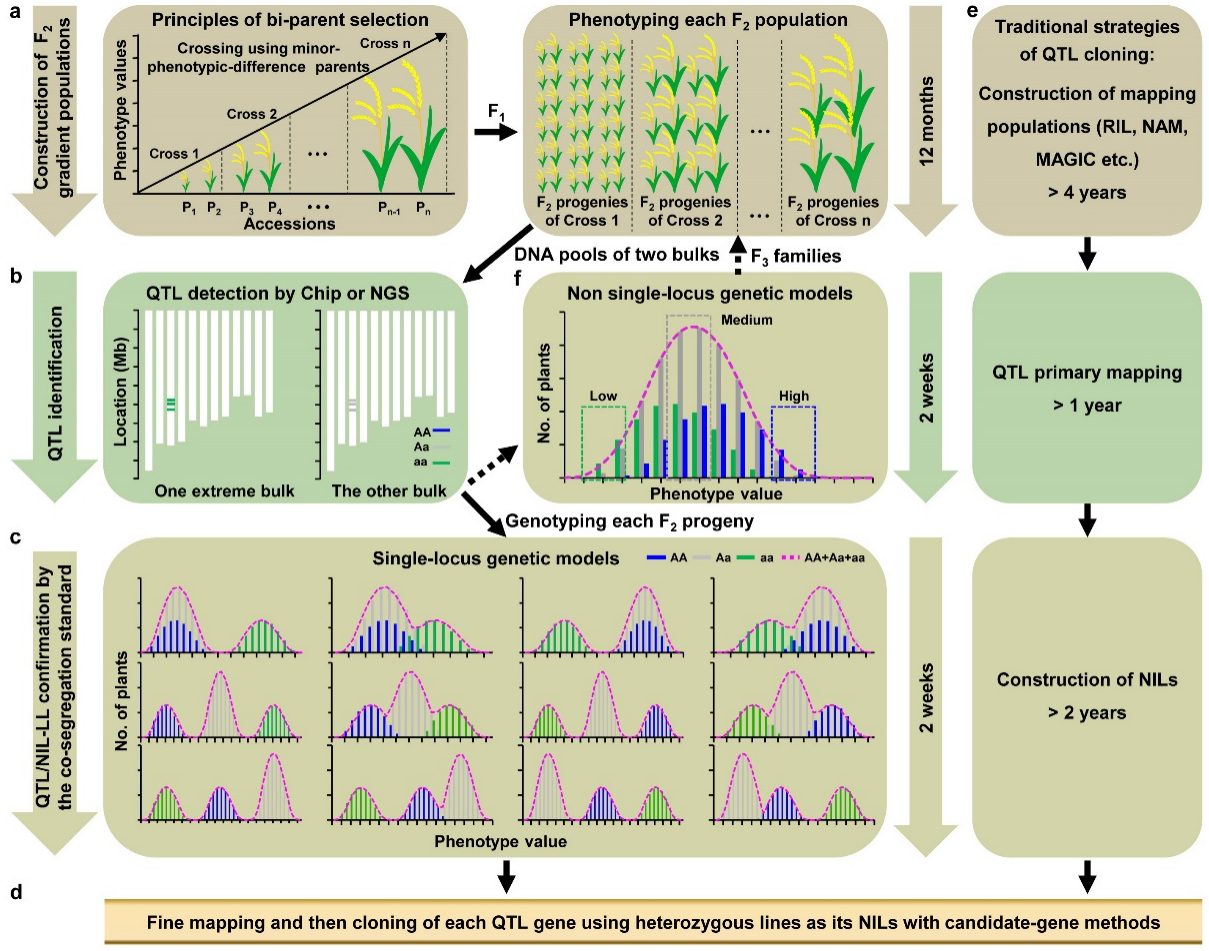
图1 RapMap定位克隆QTL基因的原理和流程
作物的产量、品质以及抗生物和非生物胁迫相关性状大多是由QTL控制的,对这些QTL进行遗传解析和分子克隆是理解遗传多样性与遗传改良作物的基础。获取单位点分离的遗传定位群体,是克隆QTL基因的先决条件。传统QTL克隆方法通过构建复杂遗传群体(RIL、MAGIC和NAM等)进行QTL定位、利用高级作图群体(NIL、SSSL和IL)实现单位点遗传,但是该过程耗时耗力,大大限制了QTL基因克隆的效率和应用进程。
为提高获取单位点遗传群体的效率,该研究首先根据显性、半显性和超显性三种遗传效应总结出了12种单位点遗传分离模型,并在此基础上提出单位点遗传分离的本质特征是在任意遗传分离群体中目标QTL/基因区段的两种纯合基因型能够与对应表型共分离,即共分离标准(图1c);该“共分离标准”是单位点遗传的充要条件,对遗传学中单位点遗传分离的传统判断标准(如双峰分布、3:1分离比等)起更正和澄清的作用,这一判断标准的更新,大大提高了获取单位点分离群体的效率和可能性。为了最大可能地满足分离群体的共分离标准,该研究提出并实践了从核心种质选取表型差异较小的双亲构建一系列F2梯度群体(F2GP)进行遗传定位的策略(图1a),以期最少化每个遗传群体中控制目标性状的分离位点数量,并克隆不同双亲组合中的主效QTL;不满足共分离标准的群体,可通过自交低、中、高值的F2或F3的若干家系,根据后代分离情况,选取目标区段杂合而背景分离位点纯合的家系来实现单位点分离。F2梯度群体结合单位点遗传的共分离标准,实现了QTL定位、验证及其类近等基因系(NIL-LL)筛选的“三位一体”的基因定位与克隆策略,大大节省了获取单位点遗传定位群体的时间和成本。该研究通过构建15个种子大小F2梯度群体,在三年内克隆了8个种子大小基因(图2),其中除了GS3、GW5、GS2、GW8和GW7/GL7等已报道基因外,还有两个新基因GL1和GW5.1及两个GS3强功能新等位基因GS3-5和GS3-6也得到克隆,这表明RapMap在QTL克隆方面具有较大潜力。

图2 粒长和粒宽F2梯度群体的亲本组合
与传统作图群体和定位方法相比,RapMap的优势主要在如下方面:一是RapMap兼具双亲本遗传群体的精度和多亲本遗传群体的遗传多样性;二是F2梯度群体实现了双亲选择和群体构建的简单性、灵活性和全面性的统一,构建时间短、成本低;三是RapMap通过共分离标准将QTL定位、QTL效应验证和类近等基因系筛选三个QTL克隆关键环节实现“三位一体”,大大提高了QTL克隆的准确度和效率,是以基因克隆为目标导向的方法;四是RapMap不仅可以克隆自然变异中的主效基因,还能鉴定克隆出自然群体中的稀有变异和微效基因;五是RapMap不依赖于复杂的软件和统计方法,而是基于基因型和表型的直接对应,避免了假阳性和假阴性的干扰,提高了QTL定位克隆的可靠性;六是通过F2梯度群体结合共分离标准,RapMap可有效地将复杂的多基因遗传降低到简单的单位点遗传,从而实现一个群体只克隆一个主效QTL基因、多个群体批量克隆多个QTL基因的突破。以上特点表明,以梯度遗传群体的构建和单位点遗传的“共分离标准”为核心的RapMap方法,是一个集QTL定位、验证及其类近等基因系筛选“三位一体”的快速、高通量基因克隆策略;该方法同样也适用于其他任何易于杂交和繁衍足够杂交后代的植物和动物的任意性状,或将成为QTL基因定位克隆的通用与首选方法,将助力功能基因组学与遗传学研究。
RapMap克隆得到的8个种子大小基因可以解释77.2%的粒形变异,对粒长、粒宽和长宽比表型预测的准确度可达0.82、0.79和0.87,表明这些基因对粒形表型具有高的贡献率和代表性,为群体水平分析粒形驯化提供了很好的前提。通过研究这8个基因在野生稻、农家种和栽培品种的等位基因频率变化,发现长粒和细粒等位基因在驯化过程中是逐步累积的,粒长和粒宽平均值也相应地变长和变窄(图3a-c),表明水稻粒形在驯化和改良进程中发生了定向选择,且这种定向选择的强度在籼稻中比粳稻中更强。同时,该定向选择伴随着核苷酸多样性降低的趋势,表明在水稻驯化改良过程中,细长粒等位基因被偏好性地选择和富集了。选择清除分析表明主效基因GS3、GW5及GW7/GL7受到强烈驯化选择,暗示了基因效应和选择压力的相关性(图3d)。相关性分析进一步表明,DNA变异、选择压力、表型变异解释率及表型关联显著性之间存在显著正相关(图3e),这说明对种子大小基因的选择压力让人类有更多机会在自然群体中鉴定到大效应的基因,比如通过全基因组关联分析(GWAS)等方法;而GWAS对微效基因或稀有变异检测能力的不足,可通过RapMap方法来进行补充。
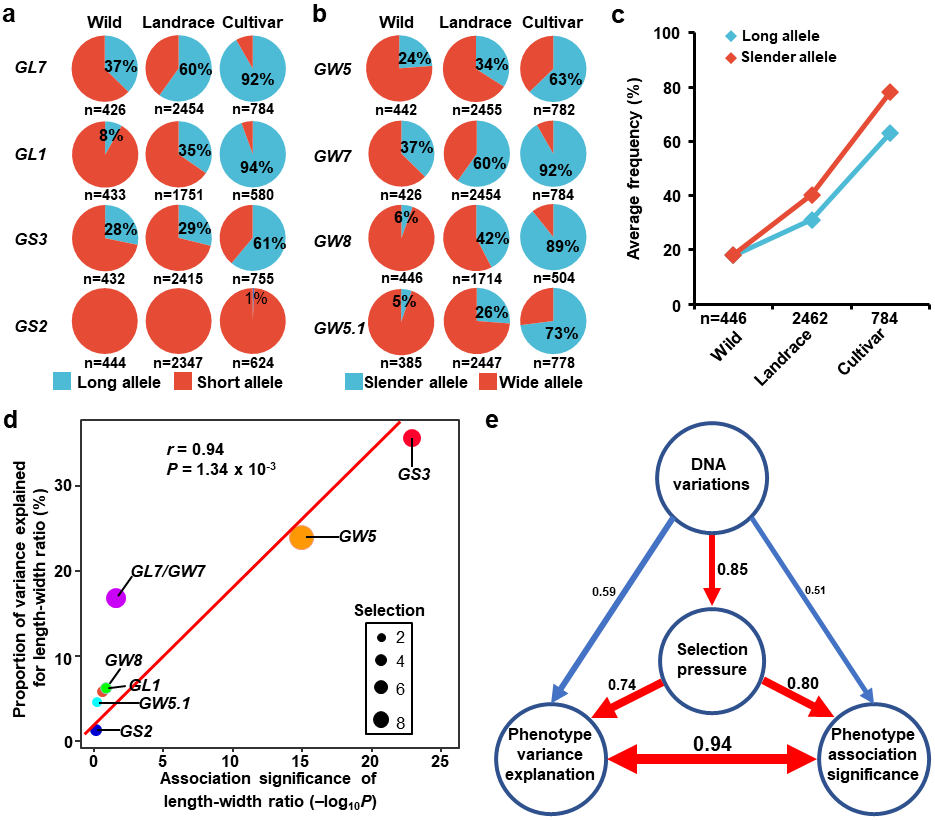
图3 八个粒形基因的定向选择以及DNA变异、选择压力、表型变异解释率及表型间的显著性相关分析
综上表明,RapMap为复杂性状QTL基因定位克隆和遗传基础解析提供了有效手段,提出了单位点遗传的本质判断标准,该研究发现的新基因和新等位基因为种子大小遗传改良提供了新基因资源;种子大小基因的选择清除分析及籼稻粒形在驯化改良过程中的定向选择等结果为水稻粒形研究提供了新见解。
我校生命科学技术学院博士后张俊成、博士研究生张德建、博士研究生樊亚伟为文章的并列第一作者,李一博课题组其他7名博士生和7名硕士生均参与了该工作,李一博教授为文章通讯作者。水稻团队练兴明教授和中科院分子植物科学卓越创新中心韩斌院士团队为本研究提供了野生稻、农家种和栽培稻的材料和数据。该研究得到了国家重点研发计划、国家自然科学基金、华中农业大学自主创新基金的资助。
【Abstract】
Cloning quantitative trait locus (QTL) is time consuming and laborious, which hinders the understanding of natural variation and genetic diversity. Here, we introduce RapMap, a method for rapid multi-QTL mapping by employing F2 gradient populations (F2GPs) constructed by minor-phenotypic-difference accessions. The co-segregation standard of the single-locus genetic models ensures simultaneous integration of a three-in-one framework in RapMap i.e. detecting a real QTL, confirming its effect, and obtaining its near-isogenic line-like line (NIL-LL). We demonstrate the feasibility of RapMap by cloning eight rice grain-size genes using 15 F2GPs in three years. These genes explain a total of 75% of grain shape variation. Allele frequency analysis of these genes using a large germplasm collection reveals directional selection of the slender and long grains in indicarice domestication. In addition, major grain-size genes have been strongly selected during rice domestication. We think application of RapMap in crops will accelerate gene discovery and genomic breeding.
论文链接:https://www.nature.com/articles/s41467-021-25961-1
审核人:李一博

