
Nature Genetics封面报道
南湖新闻网讯(通讯员 王茂军)近日,作物遗传改良全国重点实验室、湖北洪山实验室张献龙教授团队在《自然—遗传》杂志(Nature Genetics)发表题为“Genomic innovation and regulatory rewiring during evolution of the cotton genus Gossypium”的研究长文,通过棉属泛基因组和泛三维基因组研究阐明了转座子驱动基因组大小变化和染色质高级结构重塑之间的演化关系,并结合群体遗传研究揭开了纤维品质演化的基因组基础,为利用野生棉花种质资源改良栽培种提供了理论支撑。
棉花是重要的经济作物之一,是植物纤维、油料和蛋白的重要来源。棉属(Gossypium spp.)已经发现有52个种,其基因组包括A、B、C、D、E、F、G、K共8个二倍体类型(45个种)和1个AD异源四倍体类型(7个种),广泛分布于美洲、亚洲、非洲、澳洲。不同棉种植株表型差异明显,包括多年生高大树状、小灌木或匍匐生长;大部分纤维粗短,紧紧附着在种子上,只有 A 基因组二倍体和AD基因组类型的四倍体被驯化为一年生栽培种,其余棉种皆为野生种。由于长期的驯化和育种选择,栽培种棉花的遗传多态性逐渐降低,亟需从野生棉中鉴定未被利用的优异基因,拓宽育种可用的基因资源。厘清棉属基因组结构演化与纤维性状形成的关系,对于另辟蹊径提升棉花纤维品质至关重要。
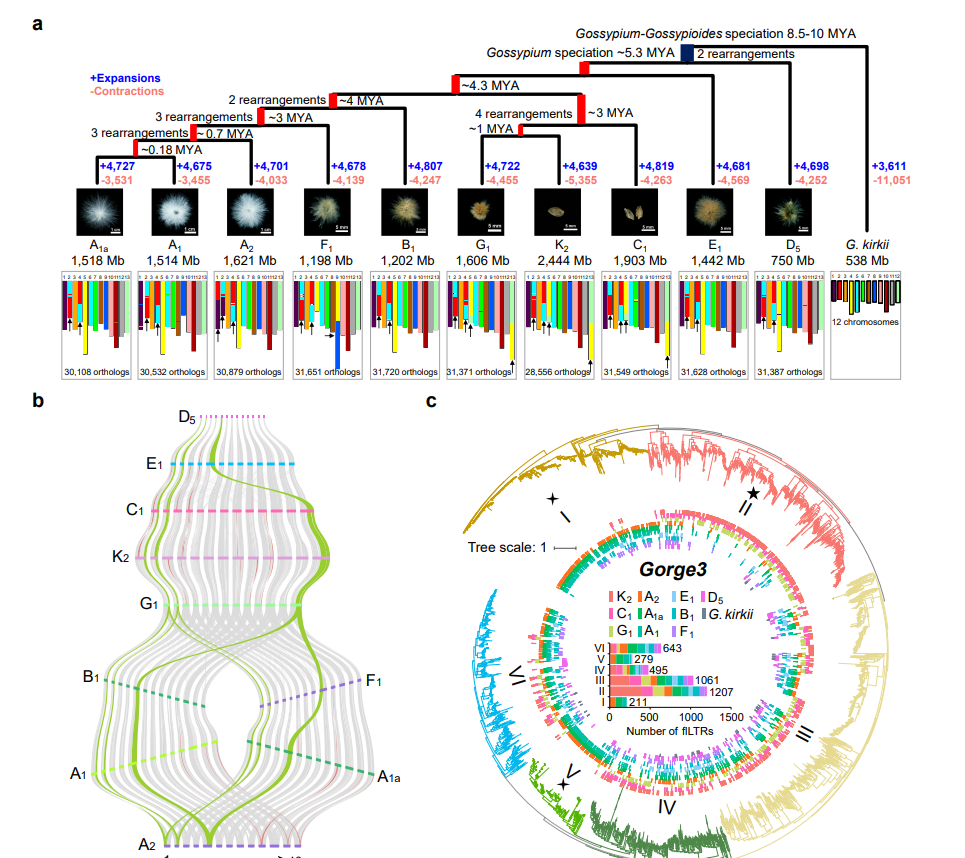
图1. 棉属系统演化和转座子家族扩增
本研究结合多种基因组学技术构建了棉属泛基因组(pan-genome),所用棉花材料包括7个代表性二倍体野生棉种和3个A基因组类型的棉花。通过不同棉种基因组的比较,阐明了棉属的演化历史:所有二倍体棉种在5百万年前(MYA)开始发生物种分化,A基因组与B1、F1基因组棉花分化时间更近,G1、K2和C1基因组棉花与A、B1、F1基因组棉花在4.3 MYA分化,E1和D5基因组棉花形成了相对独立的进化分支。通过与棉花的近缘物种叉柱棉(Gossypioides kirkii)进行比较分析,发现棉属与叉柱棉存在2–4次染色体重排事件,重排位置发生在着丝粒附近。同时,发现A、B、F等基因组间存在广泛的基因流,并发现所有棉种在末次盛冰期(Last Glacial Maximum)发生了种群规模的缩小事件。此外,研究发现转座子元件在棉花与叉柱棉分化后,发生了大规模的扩增,导致了基因组大小的不均等扩增,揭示了为什么棉属各基因组呈现至少三倍大小的变化(图1)。
为了研究不同棉种演化过程中基因组结构变异对同源基因转录调控分歧的影响,该研究构建了棉属的泛三维基因组(pan-3D genome)图谱。发现不同基因组结构变异(插入或缺失)导致拓扑相关结构域(TAD)发生重组(新的TAD形成或TAD融合),棉种特异的TAD边界形成更多地发生在不活跃的染色质区域,与棉种特异的转座子插入相关。同时,发现不同棉种间基因与基因之间的染色质互作比非编码调控区与基因之间的互作更保守,发现结构变异、转座子插入和染色质开放程度变化驱动了非编码调控元件与同源基因之间调控网络的演化,为棉花中非编码调控区的结构和功能研究提供了丰富资源(图2)。
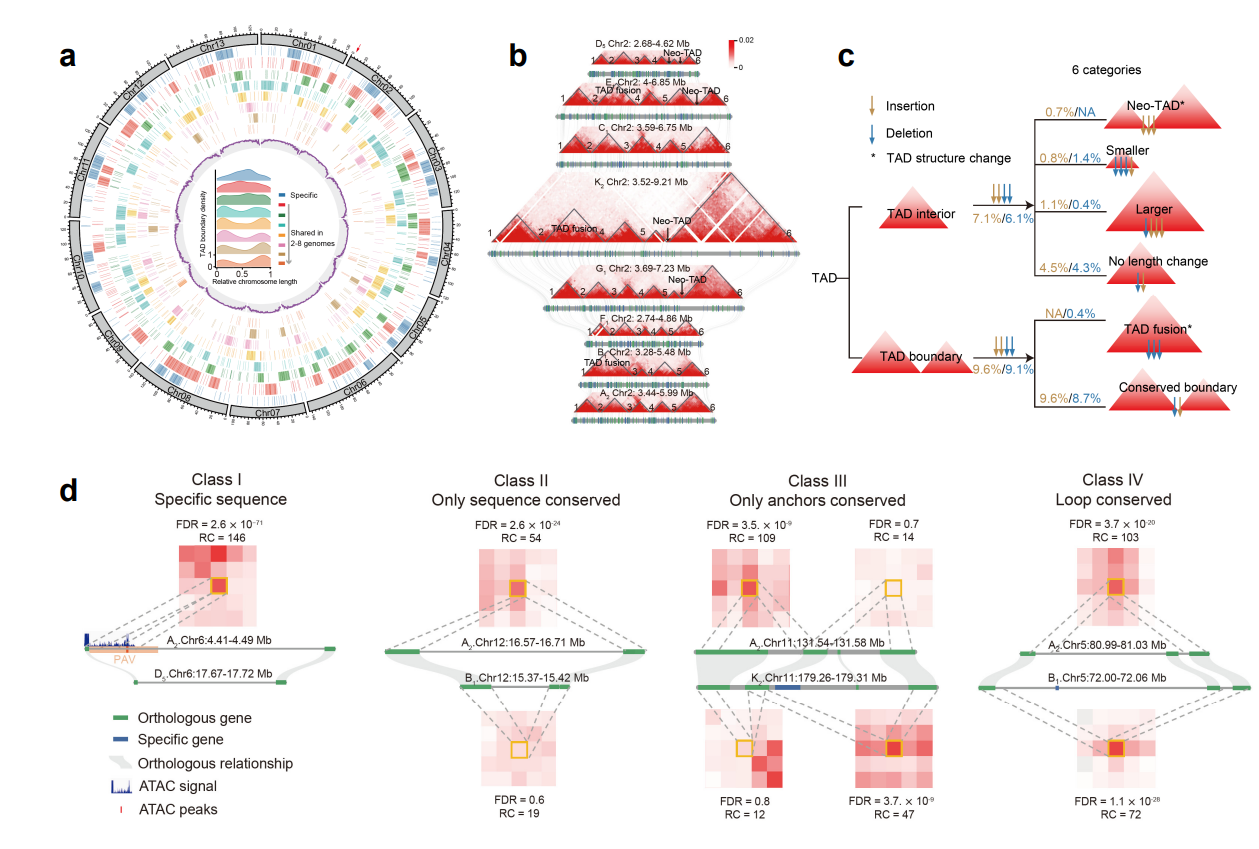
图2. 棉属泛三维基因组图谱构建与转录调控研究
进一步,研究发现基因组结构变异对纤维品质性状有影响。该研究对216份亚洲棉材料组成的群体进行纤维发育5个时间点的1005份转录组进行研究,鉴定了引起材料间基因差异表达的遗传调控位点(eQTL),解析了纤维长度变化的遗传基础。通过整合泛基因组、泛三维基因组、群体遗传等数据,揭开了棉花纤维性状从“从无到有”演化的基因组基础,探讨了纤维伸长相关基因在不同棉种中的序列变异和表达模式变化,发现SGNH基因的拷贝数变异和调控演化与不同棉种纤维品质的差异相关(图3)。
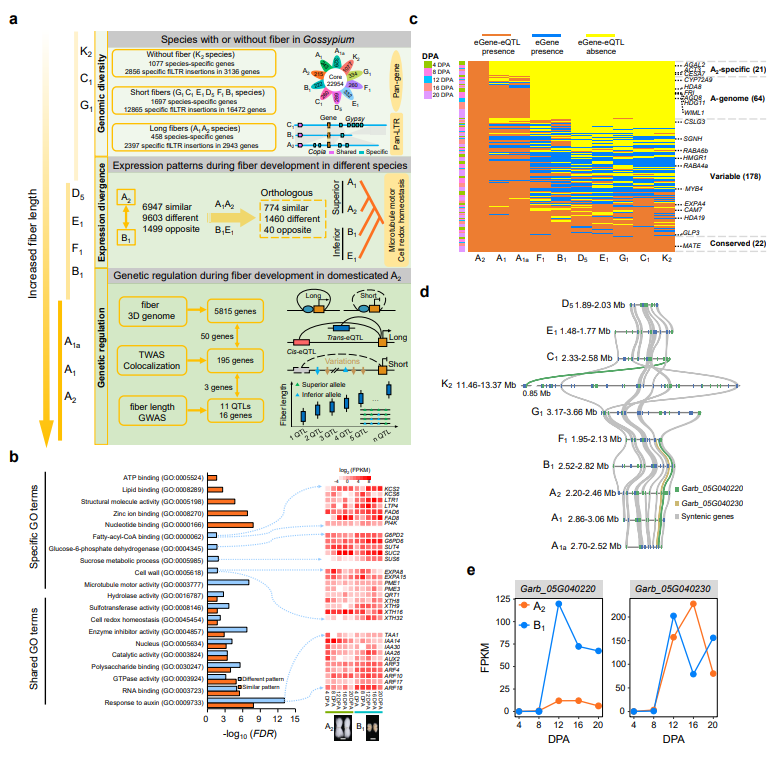
图3. 棉花纤维品质演化的基因组基础
这项研究构建了首个覆盖所有二倍体棉花基因组类型的泛基因组和泛三维基因组图谱,揭开了棉花种质资源多样性形成的基因组结构演化特征,推动了棉花纤维品质等性状演化的遗传机制研究。该研究鉴定了野生棉中可供棉花育种利用的优质基因,将助力破解优质棉育种的瓶颈问题。研究也为其他植物种属水平的种质资源演化和性状形成机制研究提供了参考。
华中农业大学作物遗传改良全国重点实验室和湖北洪山实验室的王茂军教授、博士后李健英、博士生戚正阳和本科生龙跃轩为论文共同第一作者,张献龙教授和美国爱荷华州立大学的Jonathan F. Wendel教授为论文通讯作者。中国农业科学院棉花研究研究所杜雄明研究员、美国农业部南方研究中心的David D. Fang研究员、浙江理工大学的孙玉强教授等参与了本项研究工作。该研究得到了国家重点研发计划、国家自然科学基金、湖北洪山实验室重大项目等项目的资助。
据了解,华中农业大学棉花遗传改良创新团队针对棉花生产中的重要问题,几十年来一直将棉花基因组结构和功能的基础研究和新品种培育与推广相结合,努力推动实现棉花种业科技自立自强、种源自主可控。在前期研究中,构建了栽培种陆地棉和海岛棉的参考基因组和泛基因组(Nature Genetics, 2019; Genome Biology, 2021),解析了棉花亚基因组不对称选择促进纤维驯化的遗传基础(Nature Genetics, 2017),揭开了棉花亚基因组染色质高级结构的演化特征及对纤维发育的动态协同调控作用(Nature Plants, 2018; Genome Biology, 2022),并通过远缘杂交等方法创制了棉花新种质2300余份,审定棉花新品种23个,推动了我国棉花产业的可持续发展。
英文摘要:Phenotypic diversity and evolutionary innovation ultimately trace to variation in genomic sequence and rewiring of regulatory networks. Here, we constructed a pan-genome of the Gossypium genus using ten representative diploid genomes. We document the genomic evolutionary history and the impact of lineage-specific transposon amplification on differential genome composition. The pan-3D genome reveals evolutionary connections between transposon-driven genome size variation and both higher-order chromatin structure reorganization and the rewiring of chromatin interactome. We linked changes in chromatin structures to phenotypic differences in cotton fiber and identified regulatory variations that decode the genetic basis of fiber length, the latter enabled by sequencing 1,005 transcriptomes during fiber development. We showcase how pan-genomic, pan-3D genomic and genetic regulatory data serve as a resource for delineating the evolutionary basis of spinnable cotton fiber. Our work provides insights into the evolution of genome organization and regulation and will inform cotton improvement by enabling regulome-based approaches.
在线论文链接:
https://www.nature.com/articles/s41588-022-01237-2
审核人:张献龙

